
作为一名生物汪,我们在研究基因的功能时,逃脱不掉的就是我们为了我们的实验目的,可能需要构建各种载体,比如表达载体,克隆载体,基因打靶载体等等。很多人在构建载体的时候都感觉很困难,特别是对于一个刚进入实验室的新人来说那更是难上加难,完全摸不着头脑。
那是因为其实构建载体是一个不小的工作量,需要经历很多的过程,比如首先我们需要设计引物引入酶切位点,接下来还需要经历酶切酶连等过程,只要其中的某一环节出现问题,那最终都会导致实验失败。
我认为构建载体最关键的一步就是设计引物引入酶切位点,一旦引物设计好,那接下来就非常简单了,今天重点就来教教大家如何利用snapgene轻松获得构建载体所需的引物序列。
1.打开软件,选择第一个(红线标记),然后输入基因的CCDS序列(基因的CCDS序列可在NCBI数据库中获得)

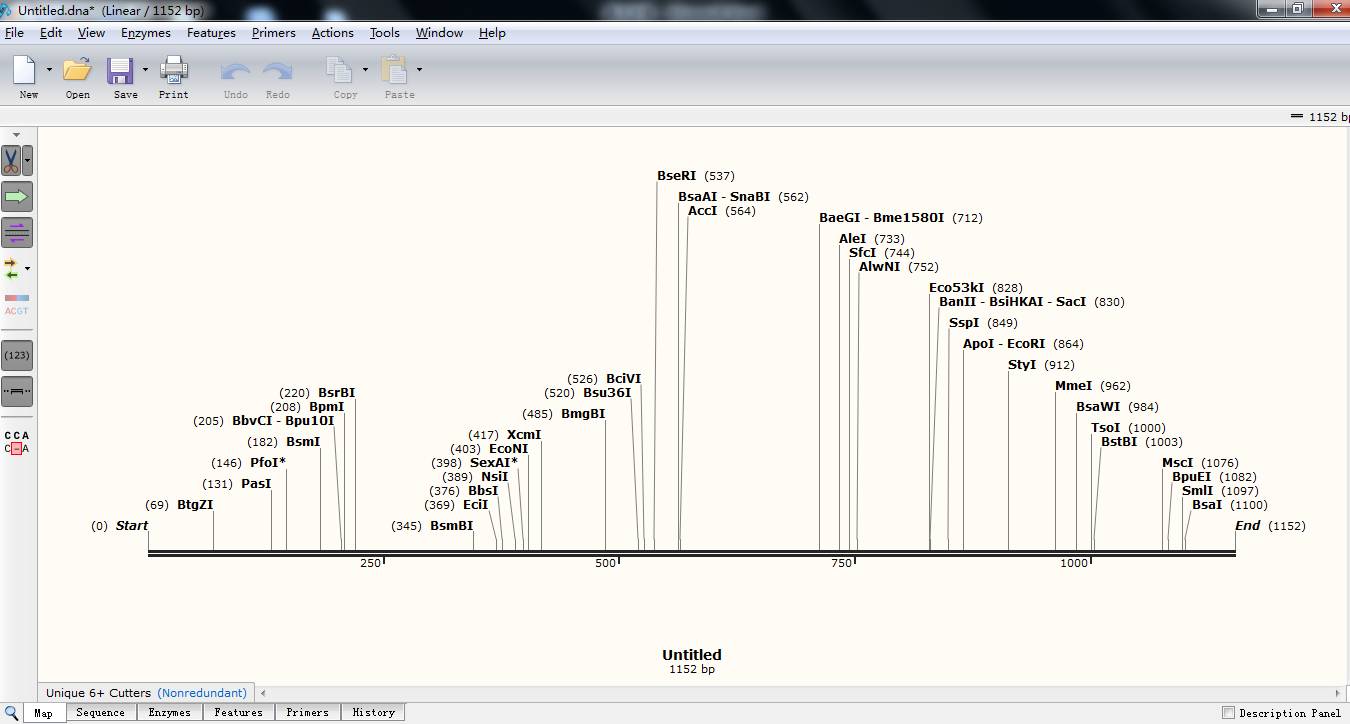
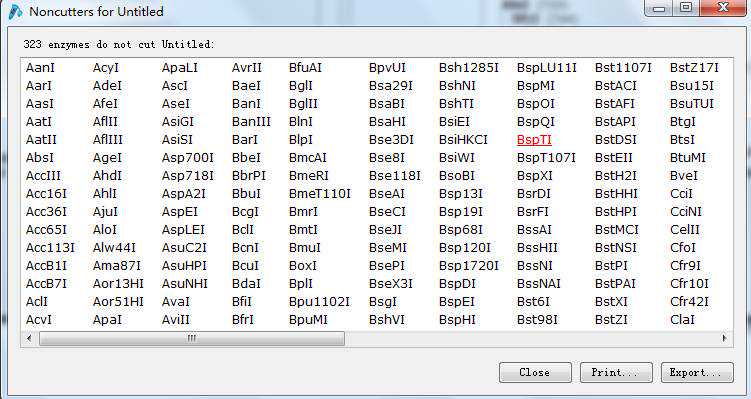
4.确定了可以用的酶后,接下来需要确定设计引物时用的这两种酶,确定方法:这两种酶最好不要邻近,最好使用双酶切有共同buffer的酶,两个酶切位点最好不要是同尾酶(切下来的残基不要互补),否则效果相当于单酶切。
5.确定了所要用的酶之后,接下来需要做的就是设计引物并引入酶切位点(注意:一定要看清楚载体上promoter的方向,将CDS正向插入到promoter后面)

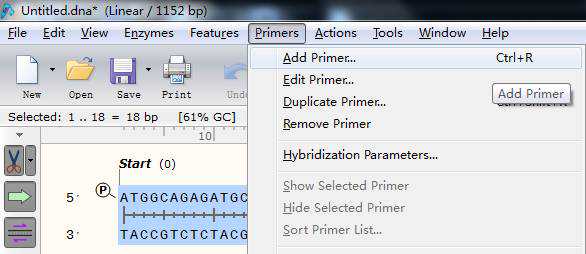
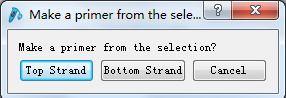
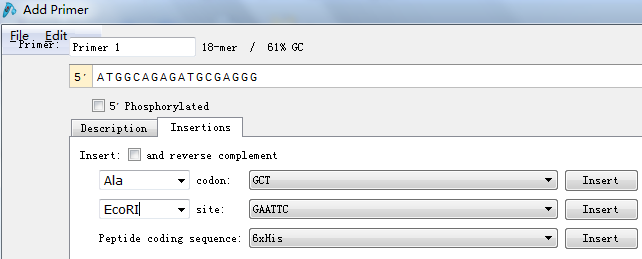
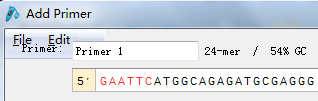
9.接下来需要添加保护碱基,我一般所有的酶的保护碱基都加三个,加哪个碱基没有要求,使GC含量及Tm值适中即可。
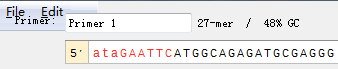
10.这样上游引物就设计好了,接下来需要检测一下引物是否会形成发卡结构,可以应用OligoCalc(),如果发现会形成发卡结构,那就需要对引物序列做出一些调整,直到发卡结构消失。
11.接下来拉取基因的下游的一部分序列,用同样的方法设计好下游引物,有一点需要注意的是,设计下游引物的时候选择bottomstrand。
这样构建载体所需的引物就设计好了,怎么样,是不是感觉其实也没有那么难,接下来就可以构建属于你自己的载体了。
欢迎投稿
tougao@
合作



