做完转录组后,我们会获得很多基因的转录本序列,如何分析这些转录本序列的开放阅读框(ORF),然后找到对应的蛋白呢?今天就为大家介绍一下使用频率最高的软件,NCBI的ORFFinder,以及另一款可以进行批量分析的软件getORF。
1.ORFFinderORFFinder是NCBI开发的一个在线分析转录本序列ORF的工具,与NCBI整合地很好。在百度里面搜索即可得到:
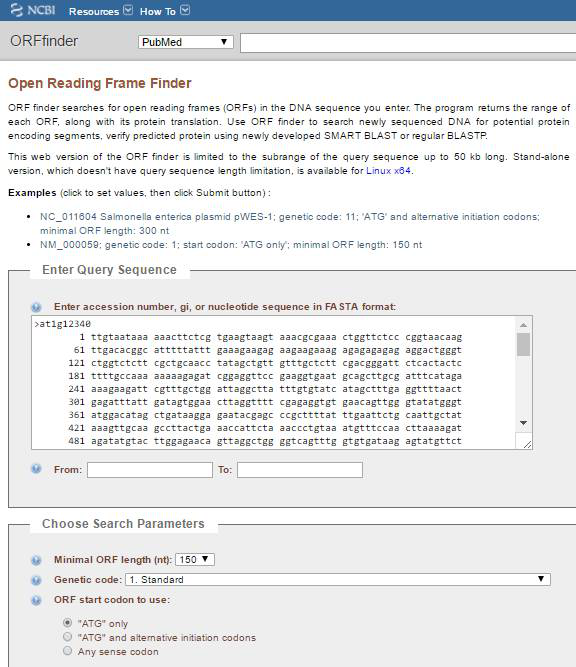
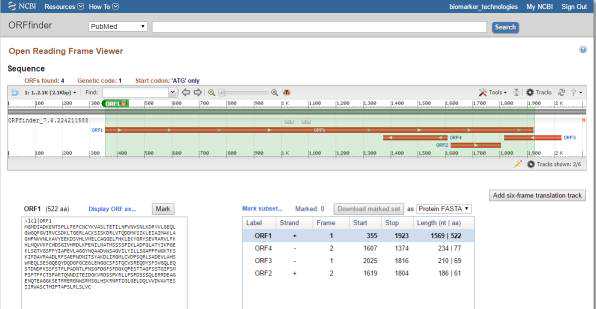
最终得到的ORF序列以图形的形式展现,可以很直观地比较ORF的位置和长度,如下:
☼
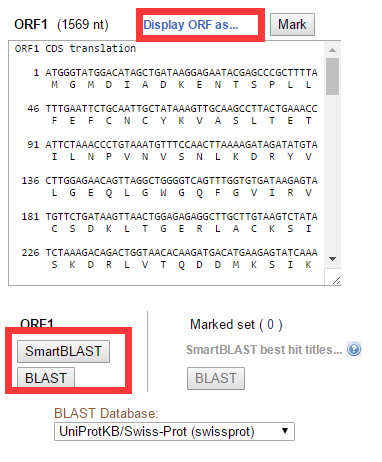
点评NCBI的ORFFinder简单易用,结果的展现形式也很直观,还能直接对得到的氨基酸序列进行blastp分析,以验证ORF的可靠性。但缺点是不能进行批量处理,只能一个一个转录本序列进行分析,这对于手上握有大量数据的同学,就有点慢了,下面介绍一款可以批量进行ORF预测的软件。
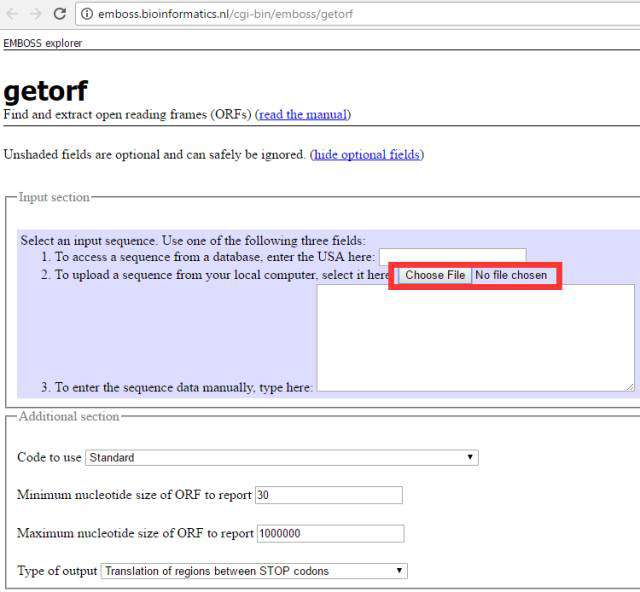
2.getORFgetORF也是一款经典的用于分析转录本序列ORF的软件,属于EMBOSS软件包。百度搜索一下getORF,会出现多个条目,是不同的科研单位搭建的在线运行getORF的平台,都可以用,小编在这里推荐一个速度比较快的平台,界面如下:
getORF与ORFFinder最大的不同就是可以上传fasta格式存储的序列(不知道什么是fasta格式的同学请自行百度),这样就可以一次上传很多条转录本序列,批量进行处理。其它的设置与ORFFinder相同,可以设置ORF长度最大和最小的限制,也可以选择是否以ATG开头(TranslationofregionsbetweenSTARTandSTOPcodons),默认是不以ATG开头(TranslationofregionsbetweenSTOPcodons)。
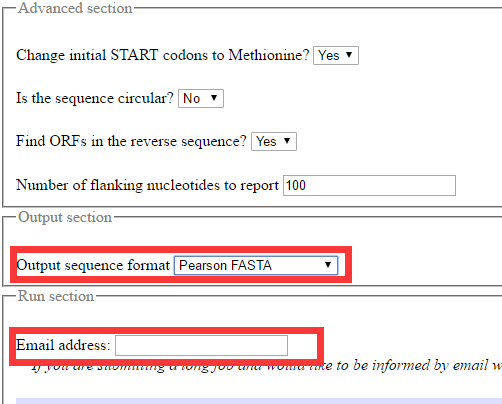
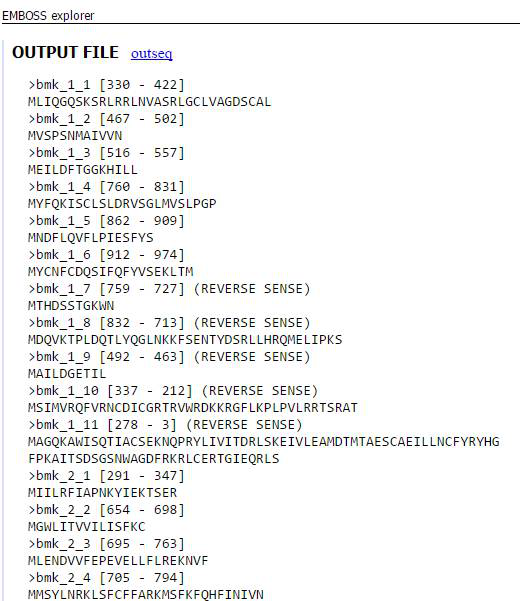
在高级设置里面还可以有多种设置,默认即可。输出的格式也可以有多种选择,还可以填写自己的email地址,会发送结果文件的链接到邮箱。最终的输出结果如下图,同一个转录本的不同ORF用后缀的数字表示(_1,_2…)。输出结果可以进行复制保存,然后提交到blastp进行分析。
☼
点评getORF最大的优势是可以进行批量处理,预测得到的ORF也很多,但界面的呈现不如ORFFinder直观。



